*해당 글은 Swin UNETR 논문 리뷰를 위한 글입니다. 궁금하신 점은 댓글로 남겨주세요.
Swin UNETR: Swin Transformers for Semantic Segmentation of Brain Tumors in MRI Images
Semantic segmentation of brain tumors is a fundamental medical image analysis task involving multiple MRI imaging modalities that can assist clinicians in diagnosing the patient and successively studying the progression of the malignant entity. In recent y
arxiv.org
Swin UNETR github: research-contributions/SwinUNETR/BRATS21 at main · Project-MONAI/research-contributions · GitHub
GitHub - Project-MONAI/research-contributions: Implementations of recent research prototypes/demonstrations using MONAI.
Implementations of recent research prototypes/demonstrations using MONAI. - GitHub - Project-MONAI/research-contributions: Implementations of recent research prototypes/demonstrations using MONAI.
github.com
Contents
2. Background Knowledge: Swin-Transformer, 3D U-Net
Simple Introduction

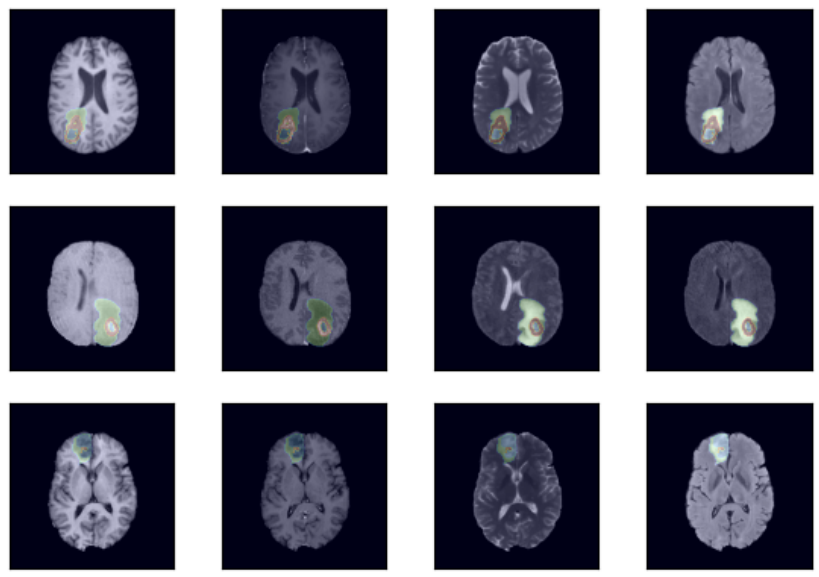
기존의 medical 분야에서는 역시나 Transformer + UNet 구조를 활용하여 segmentation 작업했다.
그러나 Transformer를 이용하면, ViT를 이용하게 되는데, ViT는 세밀한 segmentation 작업에서 단점이 있다.
따라서, Swin-Transformer 구조를 이용해서 작은 patch에서 큰 patch로 연산하는 과정을 통해서 segmentation이 더욱 세밀하고 best performance를 얻어내었다.
Swin-Transformer와 U-Net을 어떻게 결합하였을지 한번 살펴보자!
Background Knowledge: Swin-Transformer, 3D U-Net
Swin Transformer 논문 리뷰: https://kyujinpy.tistory.com/14
[Swin Transformer 논문 리뷰] - Swin Transformer: Hierarchical Vision Transformer using Shifted Windows
*Swin Transformer 논문 리뷰를 위한 글이고, 질문이 있으시다면 언제든지 댓글로 남겨주세요! Swin Transformer 논문: [2103.14030] Swin Transformer: Hierarchical Vision Transformer using Shifted Windows (arxiv.org) Swin Transforme
kyujinpy.tistory.com
3D U-Net 논문 리뷰: https://kyujinpy.tistory.com/9
[(3D) U-Net 논문 리뷰] - 3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation
*U-Net 논문 리뷰를 위한 글이고, 질문이 있으시다면 언제든지 댓글로 남겨주세요! 3D U-Net paper: [1606.06650] 3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation (arxiv.org) 3D U-Net: Learning Dense Volumetric
kyujinpy.tistory.com
해당 글은 Swin-Transformer와 U-Net의 구조를 알고 계셔야 이해하기 쉽습니다.
기존 UNETR(Transformer + UNet)의 구조에서 Swin-Transformer를 이용한 모델입니다!
해당 논문 리뷰는 관련 지식을 알고 있다고 가정하고 진행됩니다.
Method
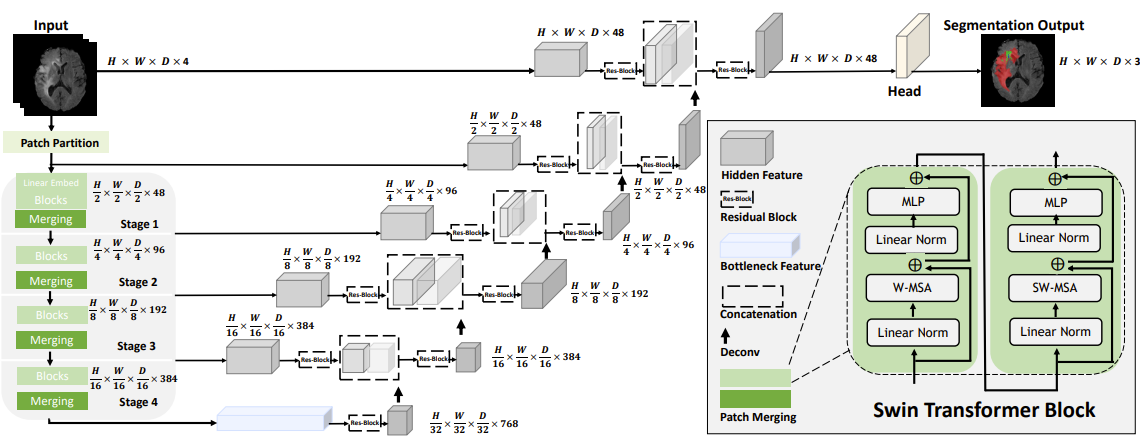
Swin UNETR의 구조는 매우 간단하다.
1. Encoder를 통해서 image의 feature map과 downsampling을 한다.
- Encoder를 통과하기 전에 Patch partition을 통해서 3D token에 대한 sequence를 만든다.
- 여기서 Swin Transformer Block을 지나치는데, W-MSA와 SW-MSA 메커니즘을 적용하는 단계이다.
- 각각의 Swin Transformer Block을 지나면서 총 4개의 중간 output을 skip-connection에 이용한다.
2. Decoder 단계에서는, 3D UNet구조를 통해서 각각의 sequence를 다시 3D voxel 형태로 reshape 후 deconvolution을 통해서 upsampling 한다.
- Skip-connection을 총 4차례 진행한 후, 원래 voxel 사이즈로 원상복귀 한다.
3. 마지막 단계에서, 기존의 input voxel의 channel을 48로 만들어 준 후, 3D UNet 구조에서 나온 최종 결과값과 concatenate를 진행한다.
4. 마지막 1x1x1 conv layer과 sigmoid를 통해서 HxWxDx3 shape으로 만들어 준다.
+) MRI dataset은 channel이 4라서 HxWxDx4 shape 이다.
+) Result의 channel 3은 segmentation 대상이 3개라서 그런 것이다. (만약 binary이면 1로 설정해도 됨.)
Swin-Transformer 간략한 용어 정리: W-MSA, SW-MSA
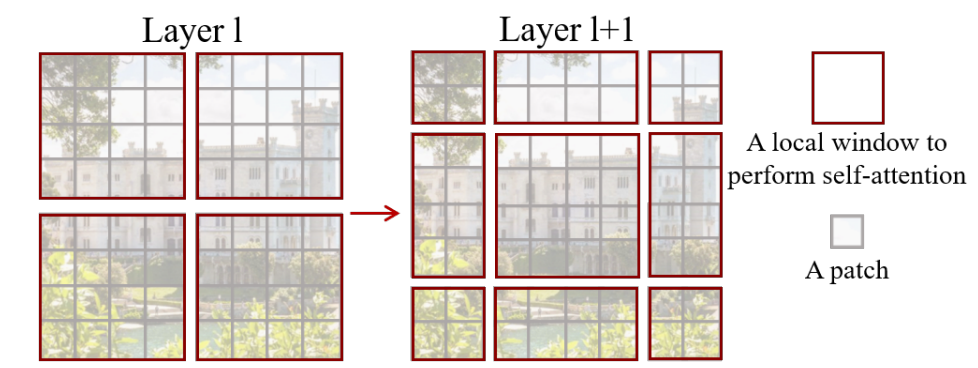
+) W-MSA는 window안의 patch끼리의 연관성이 높다고 생각해서, window 안에서만 self-attention을 하는 것을 얘기한다.

+) SW-MSA는 shift mechanism을 이용해서, window를 재배치 한 후, 재배치한 곳의 patch들은 전부 masking을 한 후에 self-attention을 하는 방법이다.
Furthermore
SSL Swin UNETR 논문 리뷰: https://kyujinpy.tistory.com/53
[SSL Swin UNETR 논문 리뷰] - Self-Supervised Pre-Training of Swin Transformersfor 3D Medical Image Analysis
*Self-Supervised learning을 이용한 Swin UNETR 논문 리뷰 글입니다. 궁금하신 점은 댓글로 남겨주세요! SSL Swin UNETR paper: [2111.14791] Self-Supervised Pre-Training of Swin Transformers for 3D Medical Image Analysis (arxiv.org) Self
kyujinpy.tistory.com
Swin UNETR에서 SSL(self-supervised learning)을 합쳐서 만든 모델이다.
한번 관심있으면 읽어보시길 추천한다!
- 2023.01.16 Kyujinpy 작성.



