*U-Net 논문 리뷰를 위한 글이고, 질문이 있으시다면 언제든지 댓글로 남겨주세요!
3D U-Net paper: [1606.06650] 3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation (arxiv.org)
3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation
This paper introduces a network for volumetric segmentation that learns from sparsely annotated volumetric images. We outline two attractive use cases of this method: (1) In a semi-automated setup, the user annotates some slices in the volume to be segment
arxiv.org
Contents
Simple Introduction

기존의 U-Net은 2D 구조였다.
즉, 2D인 이미지에 대해서 segmentation이 가능했다.
그러나 요즘 생명과 같은 biology 분야에서 segmentation을 하는 시도가 많아졌다.
Lung, Liver, Brain, Tumor 등등 특정 부위를 segmentation을 할려고 하는데, 여기서 가장 중요한 것이 의료데이터는 3차원 구조이다.
더 엄밀하게 말하면 기존의 이미지가 (H, W, 3)인 차원이라면, 의료데이터는 (H, W, S, 1)이다.
여기서 S는 slice의 약자이고, 이러한 3D 구조의 데이터는 pixel이 아닌 voxel의 개념을 갖는다.
Pixel는 1x1인 기본 unit 사각형이고, voxel은 1x1x1인 기본 unit이다.
위와 같은 3차원 데이터 뿐만 아니라, 여러 곳에서 3차원 데이터를 segmentation을 할 때 가장 기본이 되는 구조가 바로 3D U-Net이다.
모델을 한번 분석해보자.
Method

아마 내 논문 리뷰 블로그 중에서 가장 쉬운 글이 아닐까 생각한다.
기존의 U-Net 구조를 익히고 있다면 엄청 쉽다.
U-Net은 목적이 segmentation이기 때문에, input dimenstion과 output dimension이 일치해야한다.
+) 반드시 일치하지 않고 어느 정도 resize해도 되긴 하지만, 정석은 same dimension이다.
따라서 CNN기반의 U-Net은 어쩔 수 없이 CNN을 통과하면서 feature map의 크기가 줄어들기 때문에 이것을 다시 high-resolution으로 만들어줘야 한다.
U-Net의 단계는 다음과 같다.
1. Input data를 convolution 연산과 batch Normalization, 그리고 ReLU를 적용한다.
- 이 과정에서는 feature map의 크기는 줄지 않고, channel의 개수가 늘어난다.
2. 그리고 Max pooling을 적용해서 feature map의 사이즈를 줄인다.
3. 위의 과정을 N번 반복한다.
- 각 과정마다 나온 output을 모두 고려하기 때문에 그대로 둔다(?).
4. N번 반복한 최종 결과를 Up-convolution 시킨다.
- 여기서 이용되는 기법은, Upsampling으로 ConvTranspose(Deconvolution)이라고 한다.
5. Upsampling 시켜서 나온 결과는 N-1번째 결과의 dimension과 일치한다.
6. N-1번째 결과와 Upsampling 과정의 channel을 서로 연결 시킨 후, conv+BN+ReLU+Upsampling 연산을 반복한다.
- 여기서 연결되는 것을 skip-connection 이라고 한다!!
7. 최종적으로, dimension을 원상복귀 시킨 후, channel을 동일하게 만들어서 결과물을 얻는다.
+) ConvTranspose연산이다. Pytorch 자료를 가져왔으며, 3D, 2D, 1D 전부 있다.
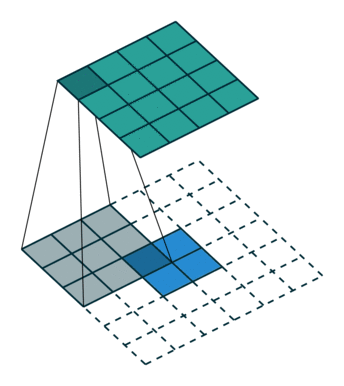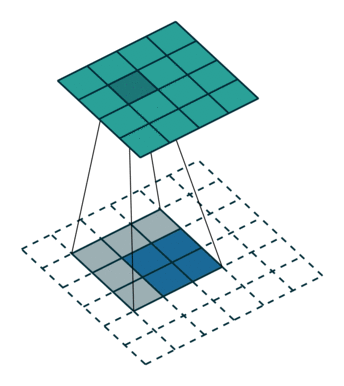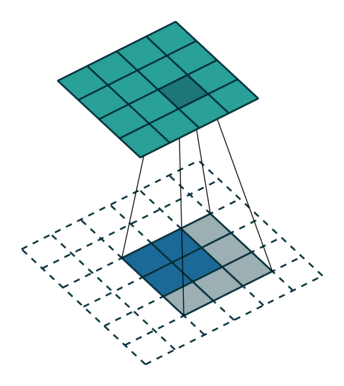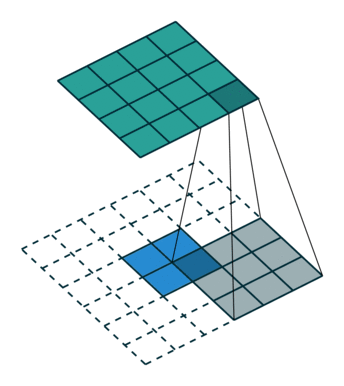
+) ConvTranspose 연산은 기존의 feature map에 패딩을 씌운 후, 이것을 CNN 연산처럼 계산하여 dimension이 높은 feature map을 만들어내는 방식이다.
Furthermore
만약 의료 데이터의 segmentation에 더욱 관심이 있다면, Transformer + Unet 구조를 엮은 논문들을 찾아보길 바란다.
*밑에 TransUnet 논문 리뷰 글도 참고해서 입문하면 좋다(?).
TransUnet 논문 리뷰: https://kyujinpy.tistory.com/13
[TransUNet 논문 리뷰] - TransUNet: Transformers Make Strong Encoders for Medical Image Segmentation
*TransUNet 논문 리뷰를 위한 글이고, 질문이 있으시다면 언제든지 댓글로 남겨주세요! TransUNet paper: [2102.04306] TransUNet: Transformers Make Strong Encoders for Medical Image Segmentation (arxiv.org) TransUNet: Transformers Ma
kyujinpy.tistory.com
- 2022.12.29 kyujinpy 작성.



